Centromero
Il centromero è una regione ristretta del cromosoma che svolge un ruolo fondamentale nella divisione del DNA durante la divisione cellulare (mitosi e meiosi). In particolare, è la zona in cui si attaccano le fibre del fuso mitotico. Dopo l’aggancio delle fibre del fuso al centromero, le due cromatidi sorelle identiche che costituiscono il cromosoma replicato vengono tirate verso lati opposti della cellula in divisione, in modo che le due cellule figlie risultanti ricevano un DNA identico.[1]
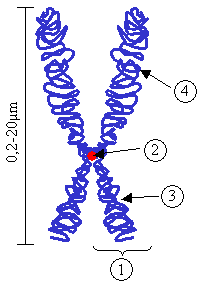
Nei cromosomi della maggior parte delle specie, il centromero occupa una posizione discreta e localizzata (cromosomi monocentrici), ma in alcune specie si estende lungo l’intera lunghezza del cromosoma (es. nei cromosomi olocentrici di C. elegans). Sebbene il loro nome sembri implicare una posizione centrale nei cromosomi (cromosomi metacentrici), i centromeri possono anche essere localizzati vicino alle estremità (cromosomi acrocentrici o telocentrici).[2]

Oltre alla variabilità nella posizione, anche la sequenza di DNA alla base dei centromeri varia considerevolmente tra le diverse specie. I centromeri “puntiformi” di S. cerevisiae e di altri lieviti si estendono per circa 125 coppie di basi (bp) e includono tre elementi di DNA conservati, la cui mutazione compromette la funzione centromerica.[2]
La maggior parte dei centromeri eucariotici è caratterizzata dalla presenza di una variante specifica dell’istone H3, che nei mammiferi prende il nome di CENP-A (Centromere Protein A). Nei centromeri degli eucarioti multicellulari, la localizzazione avviene spesso in corrispondenza o in prossimità di regioni di sequenze ripetitive, dette anche DNA satellite.[3]
I centromeri umani sono definiti da ripetizioni ricche in AT chiamate DNA alfa-satellite. L’alfa-satellite è costituito da monomeri di 171 coppie di basi (bp), disposti in tandem a formare strutture di ordine superiore (higher order arrays) che si estendono da 0,2 a 5 megabasi (Mb). Un numero specifico di monomeri forma una ripetizione di ordine superiore (HOR), che viene ripetuta centinaia o migliaia di volte.[3]
La maggior parte dei centromeri delle piante è composta da brevi sequenze ripetitive organizzate in array complessi, insieme a retrotrasposoni centromerici.[4]
Note
modifica- ^ (EN) Centromere, su www.genome.gov. URL consultato il 2 ottobre 2025.
- ^ a b Marion E Pesenti, John R Weir e Andrea Musacchio, Progress in the structural and functional characterization of kinetochores, in Current Opinion in Structural Biology, vol. 37, 1º aprile 2016, pp. 152–163, DOI:10.1016/j.sbi.2016.03.003. URL consultato il 2 ottobre 2025.
- ^ a b Kaitlin M Stimpson e Beth A Sullivan, Epigenomics of centromere assembly and function, in Current Opinion in Cell Biology, vol. 22, n. 6, 1º dicembre 2010, pp. 772–780, DOI:10.1016/j.ceb.2010.07.002. URL consultato il 2 ottobre 2025.
- ^ Shulan Fu, Zhi Gao e James Birchler, Dicentric Chromosome Formation and Epigenetics of Centromere Formation in Plants, in Journal of Genetics and Genomics, vol. 39, n. 3, 20 marzo 2012, pp. 125–130, DOI:10.1016/j.jgg.2012.01.006. URL consultato il 2 ottobre 2025.
Altri progetti
modifica- Wikizionario contiene il lemma di dizionario «centromero»
- Wikimedia Commons contiene immagini o altri file su centromero
Collegamenti esterni
modifica- (EN) centromere, su Enciclopedia Britannica, Encyclopædia Britannica, Inc.